Figure 1. 真核生物的後天免疫系統
說到 CRISPR 的發現最早可追溯到 1987 – 1993 年間,由大阪大學 (Osaka University) 的石野良純 (Yoshizumi Ishino) 1及 西班牙 University of Alicante 的 Francis Mojica 2都分別發現此特殊的序列組成持並給予 CRISPR 的命名,也是發現原核生物 CRISPR 這種後天記憶性免疫系統的開始,往後的幾年,陸續發現與其系統相關的 CRISPR array 片段、Cas9 (Cas family) 酵素、PAM (protospacer adjacent motif) 區域3以及guide RNA4等。
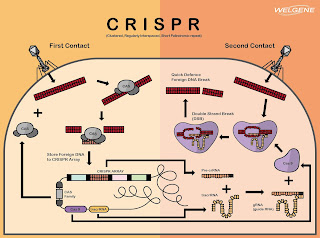
CRISPR 是古生菌及細菌中的一種後天免疫機制,當細菌被病毒感染後,病毒的雙股 DNA 會進入到細胞中,此時細胞內的 CAS 蛋白會去辨識這些外來 DNA 並進一步將其切為小片段並儲存於基因組的序列中,這些由外來序列的 spacer 和固定的 direct repeats 交互組成的區域稱為 CRISPR array,當相同的病毒第二次感染時,細胞內的 CRISPR array 會轉錄出 pre-crRNA,並和 tracrRNA (trans-activating RNA) 結合變成 guideRNA,此時細菌的 Cas9 蛋白就可以帶著 guideRNA 去辨識這些外來的序列是否和這些 CRISPR array 帶有相同的互補片段,若是相同的互補序列即可進行結合並由 Cas9 蛋白對這些外來雙股 DNA 進行切斷的 DSB (double strand break) 作用,造成這些外來 DNA 失去功能,達到快速辨識的防禦效果。
Figure 2. CRISPR 機制圖解
近幾年對細菌中 CRISPR 運作機制的逐步了解,從 2012 年開始陸續將此系統研究用在分子生物技術中,透過 CRISPR 辨識序列原則來設計出人工的 crRNA與 Cas9,運用於非細菌的 CRISPR/ Cas9 實驗,由於 Cas9 的 DSB 作用會啟動細胞體內的 DNA 修復機制如 NHEJ 或者 HDR ,因此能藉由不同的需求來將目標片段進行 indels 作用或者序列插入及互換等5 (Fig 3)。
Figure 3. 產生 DSB 後的 DNA 修復機制5。
Figure 4. Cas9 的變化型及其應用5。
Figure 5. CRISPR/ Cas9 的實際 DNA 編輯應用7。
Figure 6. 統計至2016年初,近幾年 CRISPR 相關研究於 Pubmed 有爆發性的成長8。
1. Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A (December 1987). Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology. 169 (12): 5429–33.
2. Mojica FJ, Ferrer C, Juez G, Rodríguez-Valera F (July 1995). Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning. Molecular Microbiology. 17 (1): 85–93.
3. Bolotin, A., Quinquis, B., Sorokin, A.,and Ehrlich, S.D. (2005). Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology 151, 2551–2561.
4. Brouns, S.J., Jore, M.M., Lundgren, M., Westra, E.R., Slijkhuis, R.J., Snijders, A.P., Dickman, M.J., Makarova, K.S., Koonin, E.V., van der Oost, J. (2008) Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960-964.
5. Jeffry D Sander & J Keith Joung. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology 32, 347–355 (2014) doi:10.1038/nbt.2842.
6. Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F. Science. Multiplex genome engineering using CRISPR/Cas systems. 2013 Feb 15;339(6121):819-23. doi: 10.1126/science.1231143. Epub 2013 Jan 3.
7. Francisco J. Sánchez-Rivera & Tyler Jacks. Applications of the CRISPR–Cas9 system in cancer biology. Nature Reviews Cancer 15, 387–395 (2015) doi:10.1038/nrc3950.
8. Biosynthesis, Chemically Modified Nucleic Acids for CRISPR-Cas. http://www.biosyn.com/tew/Chemically-Modified-Nucleic-Acids-for-CRISPR-Cas.aspx







留言
張貼留言