
然而 NGS 技術帶來的突破並未停歇,而是繼續由其他角度提升癌症解析層次。綜觀所有癌症檢測,醫院日常進行的臨床病理 IHC、FISH 實驗,應為數量體最龐大、驗證充足、臨床經驗豐富的經典癌症檢測,這兩個技術多用於直接檢測蛋白質的表現量,或間接評估染色體變異引發的蛋白質融合,由此可知醫院日常的癌症檢測基礎是「蛋白質」。雖然 NGS 技術未能直接測定蛋白質,但 NGS 能夠自一個樣品中定序出數億條的序列 ( hundreds of millions of reads per sample ),這樣的能力再搭配上發展成熟的目標增幅 ( target enrichment ) 技術,使癌症醫學得以處理 RNA 降解嚴重、序列多變、數量落差問題,以高解析度方式間接評估蛋白質表現量或融合,建立更全面性的 DNA translocation -- RNA fusion -- Protein fusion 生物中心法則 ( central dogma ) 連結。因此國際上的大型癌症研究計畫,例如歷時 13 年多 ( 2005-2018 ) 的 TCGA ( The Cancer Genome Atlas ) 計畫,或是 2004-2015 致力於 Glioblastoma transcriptome data ( RNA-Seq ) 的 the GLIOCAT study,皆將 NGS RNA-seq 列入數據蒐集環節,並於計畫後源源不斷找出新的連結。
若以 TCGA 計畫為例 ( 圖一 ),相關研究顯示近 16.5% 癌症的發生與基因融合 ( gene fusion ) 密切相關 1,並鑑定出 6% 案例具可用藥性,透過基因融合轉譯產物 ( RNA fusion transcripts ),直接檢測 fusion gene 轉錄 / 表現能力,比定序整個基因體 DNA 更快速找出變異,有利了解 fusion RNA expression 對癌細胞的影響。
 |
| 【圖一】Driver Fusions and Their Implications in the Development and Treatment of Human Cancers |
因 RNA fusion 對癌症醫學的重要性不言可喻,近年精準醫學檢測也開始導入 NGS RNA fusion 定序,並由臨床實證中顯示其諸多特點 2 ( Table1 ):
( 1 ) 更全面了解對癌症的影響
一次實驗可偵測所有發生 gene fusion 的區域、基因重排的變異狀況、基因異常表現情形。
( 2 ) 可用於 liquid biopsy 樣品
組織檢體難取得、不易處理,因此血液樣品中的 cell free RNA 是更方便的檢體類型。
( 3 ) 高準確度 ( sensitivity & specificity )、高通量
在成本經濟與實驗便利的條件下,讓臨床獲得更充足、有效的癌症基因訊息。
雖然有諸多優點,然而癌症 fusion RNA 基因定序存在許多困難之處,包含:
( 1 ) 樣品條件
樣品萃取是 NGS 實驗關鍵的第一步,需取得完整度足夠的 RNA 樣品,才可進行有效定序,參考 RNA 樣品品質指標 — DV200 ( RNA > 200nt 的片段比例 ),以預估 RNA 完整性,建議至少使用 DV200 ≥ 30% 之 RNA 3,當樣品品質 / 完整度越高,基因定序結果的可信度越高。
( 2 ) 資料分析
Fusion gene 變異頻率微弱,增加了突變訊號檢測的困難性,Gene junction 區域 reads 的 coverage ( 覆蓋率 ) 與 abundance ( 表現量 ) 不足會造成 RNA fusion 假陰性結果。ICGC-TCGA DREAM Somatic Mutation Calling in RNA ( SMCRNA ) 計畫利用 RNA-seq 取得大量癌症數據,提供準確預測 RNA fusion 訊息的分析方式,可透過 https://github.com/SMC-RNA-challenge 來參考分析 4。
( 3 ) 建庫方法
一般研究用的 poly-A RNA Selection 無法用於臨床降解嚴重的 RNA 樣品,並且無法將定序集中在 exon junctions 之上。RNA capture 是一種抓取 RNA CDS exome 技術,其利用探針將重要的目標基因 exons 抓取下來,可獲得更顯著的 RNA fusion / splicing 訊號。美國權威醫學中心 — 梅奧醫院 ( Mayo Clinics ) 為發展個人化醫療,使用 Agilent SureSelect 系統 ( Table2. T47D+_Capture ) 進行 RNA ( SureSelect All exon+UTR v4 ) 抓取並建庫 5,偵測到近 2 倍的 fusion reads ( Table2 ),更可能發現 mRNA 之外,其他上游基因產物 pre-mRNA 或 DNA 的 fusion 情形,找到完整的潛藏變異。
 |
| 【Table2】Fusion Supporting Reads for Sample T47D+ with and without All-Exon Capture |
 |
| 【圖二】Kinome-centred RNA sequencing identifies novel gene fusion. |
 |
| 【圖三】Multiple mutations identification: FGFR3-TACC3 translocation (A, B) and serine-to-cysteine substitution (C). |
Agilent SureSelect 系統合適癌症 RNA fusion 偵測,歸因於 SureSelect 的:
1. 基因內容多元設計彈性
包含規格型或客製化方案,如應用現有的 All Exon Target Enrichment 進行全基因掃描,有助快速發現可能的、新的致癌位點;而 Custom Panel 可自由設計最適化內容,對於各癌別基因檢測的研發或臨床更深入的研究,具有高度應用價值。
2. 樣品製備方法學最佳化
使用 Probe 抓取法進行 RNA capture 配合 MBC ( Molecular Barcode ) 設計 ( 圖四 ),幫助 transcriptome 分析可以更高的效率獲得豐富、不遺漏的基因訊息,在合適的基因範圍,加強定序深度,在有限資料中發現更多的基因複雜性。
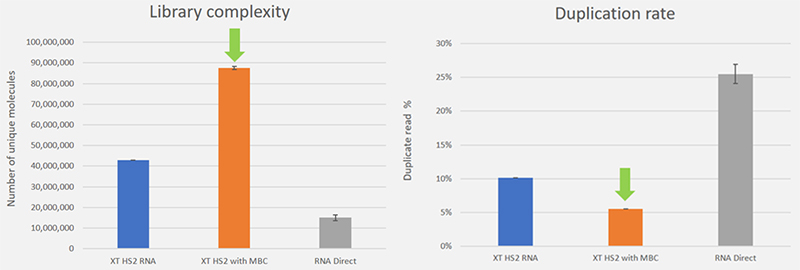 |
| 【圖四】MBC 提升文庫複雜度、減少重複讀取序列 |
3. 臨床樣品 ( FFPE ) 最適化
克服高難度的臨床樣品條件,仍保有良好製備品質 ( 高文庫複雜度 ) 與定序效能 ( 低重複序列數據 ),增加臨床研究實用性。
克服高難度的臨床樣品條件,仍保有良好製備品質 ( 高文庫複雜度 ) 與定序效能 ( 低重複序列數據 ),增加臨床研究實用性。
 |
| 【圖五】FFPE-RNA library preparation 最優化 |
NGS 成熟的技術使得「RNA fusion」成為新興的癌症偵測指標,為醫學研究、甚至臨床診斷、預後追蹤、及新的治療策略開發帶來極高的應用價值。2019年,歐洲腫瘤內科學會 ( ESMO ) 整合 gene fusion 檢測的流程 7 ( 圖六 ),為了 RNA fusion 檢測能夠再進一步發展,有效應用在癌症診斷,因此將 RNA NGS 作為第一線檢測工具,搭配 IHC 法確認癌變基因,加速臨床作業效率、節省醫療成本,特別是幫助病徵、病因不明確的病人爭取時間,可以盡早對症治療。
RNA NGS 檢測 gene fusion 的優勢在於突破 DNA 提供的遺傳訊息,發現更多細胞真實的基因異常表現狀況,如何影響癌症生成。Cancer genome projects 已進入了成熟期,而 RNA fusion 檢測才剛起步,可以預期 RNA fusion 將為新型癌症檢測的發展重點。
 |
| 【圖六】ESMO Translational Research and Precision Medicine Working Group recommendations. |

留言
張貼留言