在基因診斷領域,短讀長定序(short-read sequencing, SRS)雖能高效檢測單核苷酸變異(SNV)和小型插入缺失(InDel),但其侷限性在處理複雜基因組區域時日益顯現。例如,重複序列擴增(如短串聯重複STR)、假基因干擾、結構變異(SV)及表觀遺傳異常(如甲基化缺失)等,常導致高達25%的臨床樣本無法獲得明確診斷。傳統解決方案需結合多種技術(如核型分析、甲基化特異性PCR、長片段PCR等),不僅流程繁瑣,成本與時間負擔也成為臨床應用的障礙。
近年來,PacBio的HiFi長讀長定序技術(HiFi long-read sequencing, LRS)憑藉高精度(>99.9%準確率)與長讀長(平均15-20 kb)優勢,逐步展現取代多重檢測的潛力。荷蘭Radboud大學醫學中心團隊近期於《The American Journal of Human Genetics》發表的研究,首次系統性驗證HiFi LRS在複雜臨床樣本中的診斷效能。該研究以100例已知致病變異的挑戰性樣本為對象,涵蓋STR擴增、假基因區變異、複雜結構重組等145個傳統技術難以偵測的遺傳異常,結果顯示HiFi LRS不僅能自動偵測83%的變異,結合人工輔助後總偵測率更達93%,遠超越SRS的41%偵測率。此成果標誌著基因診斷邁向「單一技術整合多重變異解析」的新階段,為罕見疾病與複雜病例的分子診斷開闢更精準且高效的途徑。
研究共選取 100 個樣本,涵蓋145個已知難以透過SRS偵測的致病變異類型,包含 25 個短串聯重複 (STR) 擴增、34 個假基因區或同源基因座變異、23 個複雜結構變異,以及其他如印記區域與粒線體變異等特殊情形。其中,42 個樣本(含 70 個變異)曾納入早期使用 SRS 的研究,因此能直接對比兩種技術平台在真實臨床案例中的偵測能力。
 |
| 圖1.(A) 所有 145 個變異按變異類型分群的樣本分布 (B) 每位患者過去在診斷實驗室中所需的檢測方法種類與次數。 |
所有樣本皆使用 PacBio Revio 平台進行 HiFi 定序,預期達到 30× 的全基因體覆蓋率。DNA 抽取與文庫建製依照標準流程操作,將 gDNA 剪切至 15–18 kb 長度後建庫,經 BluePippin 選擇出 >10 kb 的片段後進行 24 小時定序。平均資料輸出為 94.0 Gb,中位覆蓋率達 29.73×,平均讀長為 15.35 kb。在分析流程上,整合官方與開源軟體,針對不同變異類型設計專屬的模組,若軟體無法偵測預期變異,則以 Integrated Genomics Viewer (IGV) 進行人工視覺化確認,確保難辨識變異不因演算法限制而遺漏。
結果顯示,在 145 個變異中,有 120 個(83%)可透過變異分析工具自動辨識,包含 61 個結構變異、20 個 STR 擴增與 40 個 SNV 或 InDel,其中STR 擴增範圍從 16 至超過 150 個重複單元不等,分布於 12 個不同基因,多數變異位於具有高度同源性的區域。當上述演算法無法自動偵測特定變異時,研究團隊也整合視覺化與人工確認機制,包括使用IGV手動檢查比對結果,或以NAHRwhals分析大型非等位重組(NAHR)事件結構,加上14個(10%)透過人工視覺輔助確認的變異,變異總偵測率達93%,僅有11個(7%)因GA-rich重複序列、X染色體甲基化比例偏移或變異斷點落於50 kb以上片段重複而無法辨識。
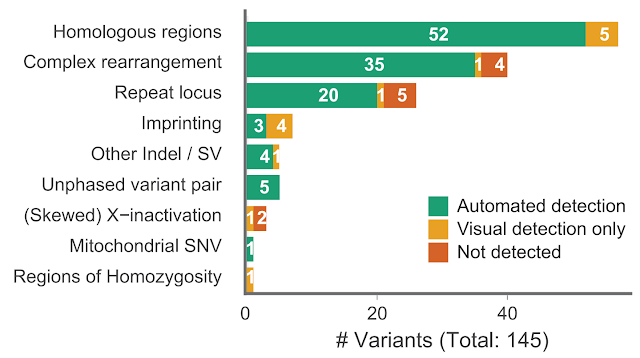 |
| 圖2.依據變異類型,顯示使用 LRS 進行自動變異偵測與人工視覺化檢查後的總偵測率。綠色表示可自動偵測(83%),橘色表示人工可視化判讀(10%),紅色為未偵測到變異(7%)。 |
研究中以多項實例顯示LRS提供了傳統技術難以處理的變異資訊,例如透過HiFiasm進行單樣本haplotype-aware的de novo組裝,成功辨識STRC缺失與其假基因STRCP1之差異,反映了LRS在解決序列高度相似區域的變異方面的優勢;搭配 pb-cpg-tools 成功分析出母系第14號染色體上的印記缺陷,源於單親異二體性(uniparental heterodisomy, UPD),此類印記缺陷通常難以透過一般WGS或WES偵測,需另行進行MLPA或methylation-specific PCR檢測,LRS讀段精準地呈現出甲基化缺失分布,成功解釋患者表現型背後的遺傳與表觀異常機制;在OPN1LW/OPN1MW(紅綠視覺色素)高度同源基因群的樣本中,藉由Paraphase分群可區分出OPN1MW2之具致病性等位拷貝,使隱性致病突變的定位與變異型別鑑定更加精準,為視覺障礙等X染色體相關疾病提供分子佐證。
 |
| 圖3.(A)針對STRC基因區域的從頭組裝結果,透過Paraphase工具將讀段按推定來源基因(功能基因或假基因)分組。(B)此圖顯示樣本P50-G3於第14號染色體印記區的CpG甲基化分佈 |
LRS不僅能偵測傳統方法難以處理的變異,亦能提供額外的結構解析能力,例如在涉及13號染色體與Y染色體不平衡轉位的樣本中,僅在採用T2T(telomere-to-telomere)完整參考基因體替代GRCh38後,才能精確識別出該不平衡轉位的斷點與接合順序;利用 Hifiasm 組裝資料與 NAHRwhals 視覺化工具,成功重建出一段橫跨 CEP85L 與 MCM9 基因,長達約200 kb的複雜基因重組,此類異常在短讀資料中極易被誤判為多個獨立 CNV 或插入/缺失事件,而LRS的長讀長使得單一讀段可涵蓋整個重組區,配合組裝結果清楚呈現不同片段的相對位置與接合點,使該複雜事件得以完整定義,這種高解析能力對於理解致病機制與個別化治療策略規劃具有高度應用價值。
 |
| 圖4. 橫跨CEP85L與MCM9基因的約200 kb複雜基因重組。 |
進一步分析11 個(7%)因技術限制而無法自動或人工識別的變異,其中重複序列富含GA鹼基(GA-rich repeats)的變異佔多數,包括FXN基因中的(GAA)n擴增,以及RFC1基因中的(AAGGG)n、(AAAGG)n和(ACAGG)n等重複序列,該類序列可能因形成非B型DNA結構(如Z-DNA或三鏈DNA)而干擾DNA聚合酶的延伸反應,進一步導致HiFi read片段的品質下降與讀長變短,甚至造成特定等位基因的覆蓋缺失(allelic dropout)。此外,X染色體失活偏斜(skewed X-inactivation)偵測結果也顯示,在30×覆蓋條件下,若偏斜程度低於80%,LRS在甲基化模式上的偵測靈敏度可能不足,未來尚需設計專屬的甲基化模組以提升靈敏度。
覆蓋率(coverage)是影響變異偵測能力與資料品質的核心因素之一,為評估覆蓋率對偵測表現的影響,研究團隊進行降採樣(downsampling)模擬。當原始177個自動偵測變異於30×覆蓋時均可辨識,降低至20×時仍可偵測96.0%,15×為89.5%,10×則降至76.8%。其中以位於假基因區的SNV、SV與重複相關CNV降幅最大,STR與非同源SNV則相對穩定。這項結果說明,15× 覆蓋可能為實務應用的最佳平衡點,但30×覆蓋對維持LRS於高複雜度區域的敏感性具關鍵意義。
 |
圖 5.降採樣實驗,呈現不同全基因體覆蓋率(10×、15×、20×、30×)下,各類型變異的自動偵測成功率變化(共 177 個呼叫)。位於同源區與重複區的變異對覆蓋率最敏感,在10×下幾乎半數無法正確呼叫,而STR與簡單SNV則在10×下仍保有超過80%偵測率。 |
在與SRS進行對比的42個樣本中,LRS達成89%的自動偵測率(62/70),遠優於Illumina SRS僅有的41%,即使SRS經過人工輔助分析後(如手動查看IGV、針對特定表型搜尋),偵測率也僅能提升至約71%,且需要耗費大量時間與專業判讀人力,仍有20個變異完全無法偵測,這些變異大多與假基因區、高度同源序列或大型結構變異有關,證明了LRS對基因體複雜區域的優勢。研究結果表明,LRS能夠有效識別各種難以透過SRS檢測到的變異,從資料層級到臨床應用層級,PacBio HiFi LRS在偵測靈敏度、變異涵蓋性、資料解讀深度以及診斷流程簡化上,表現均優於SRS定序平台。更重要的是,LRS提供了真正單一技術平台下,涵蓋「小變異、結構變異、STR擴增、假基因分辨、甲基化異常」等多重遺傳特徵的全方位解決方案,為未來罕病、複雜表型與疑難病例的分子診斷提供更精準地方法。
整體而言,此篇研究證實 PacBio HiFi 長讀定序技術能有效涵蓋幾乎所有主要類型的致病變異,並提供結構變異、序列變異、假基因區辨識、相位與甲基化分析等全面資訊。雖然目前 LRS 的成本與產能仍為限制,但其技術潛能與解析度為臨床應用打開新的應用與可擴展性。
【參考文獻】
- Ho¨ps et al., HiFi long-read genomes for difficult-to-detect, clinically relevant variants, The American Journal of Human Genetics (2025), https://doi.org/10.1016/j.ajhg.2024.12.013
留言
張貼留言